在2013年,来自麻省总医院的研究人员发现使用CRISPR-Cas RNA引导性核酸酶的一个重要局限:会在预期靶点以外的位点上生成多余的DNA突变。此后陆续有研究直接说明了CRISPR/Cas9存在严重的脱靶性,即该技术可以发生非特异性切割,引起基因组非靶向位点的突变,这样会造成研究结果的不确定性以及研究工作的大量增加,这一问题严重地限制了Cas9的应用。
因此科学家们希望能通过全基因组检测脱靶剪切,近年来研发了不少检测脱靶剪切酶活性的实验方法,比如近期Nature Methods报道的两项技术,这些技术是什么,具有何种优势,具体让我们来看看:
GUIDE-seq
以往人们在检测CRISPR-Cas核酸酶诱导的脱靶DNA断裂时,往往事先假定脱靶位点与靶标位点相似。GUIDE-seq是首个不需要这样做的方法,而且它相当灵敏 。
一组研究人员利用一种短双链寡核苷酸来标记CRISPR-Cas诱导的脱靶断裂,测序这些标签所在的基因组区域,就能够确定脱靶突变的位置。研究表明,就算某个脱靶突变的出现频率低至0.1%,GUIDE-seq也能够检测得到。由于许多脱靶突变发生在与目标位点差异很大的地方,因此脱靶DSB的数量和位置是很难预测的。
现有工具主要是通过分析目标序列来预测脱靶突变,研究人员将这些工具与GUIDE-seq进行了比较。研究显示,上述工具在预测已验证的脱靶位点时比GUIDE-seq差很多,还会错误检出实际上不被酶切的位点。此外,研究人员还对比了GUIDE-seq和ChIP-seq(检测蛋白与DNA的结合),结果证明ChIP-seq并不是鉴定 CRISPR-Cas脱靶DSB的可靠方法。
Digenome-seq
来自韩国首尔大学、首尔基础科学研究所等机构的研究人员在Nature旗下子刊《Nature Methods》发表一项研究,成功证实CRISPR-Cas9在人类细胞中有精确的打靶作用,他们开发出一种强大、敏感、无偏见和具有成本效益的方法——Digenome-seq,可在全基因组范围内检测人类细胞中的CRISPR/Cas9脱靶效应。
在这项研究中,研究人员称,虽然RNA引导的、通过CRISPR-Cas9系统的基因组编辑已经被广泛应用于生物医学研究,但是Cas9核酸酶的全基因组靶向特异性仍然是有争议的。为此,他们提出一种方法Digenome-seq,利用基因组测序查找CRISPR-Cas9可能突变产生的打靶和脱靶序列。他们用Cas9核酸酶在试管中消化人类基因组DNA,然后进行全基因组测序。这种体外消化可产生打靶和脱靶序列的独特模式,可以通过计算确定。此外,个在sgRNA末端添加组成CRISPR-Cas9的鸟嘌呤核苷酸,研究人员成功地制备了这种高度发达的可编程核酸酶,在人类基因组中它没有可测量的脱靶效应。
此后,这一研究组又公布了Digenome-seq升级版,并且他们用这一技术同时分析了11个CRISPR-Cas9核酸酶在整个基因组中的特异性,大大节省了CRISPR脱靶分析所需的时间和经费。
研究人员先将基因组DNA从人类细胞中提取出来,然后用多个sgRNA-Cas9组合进行消化,最后进行全基因组测序。他们用一种新的DNA剪切评分系统,鉴定了Cas9在基因组中的剪切模式,即打靶和脱靶位点的特性。研究表明,转录自双链寡核苷酸的sgRNA引起了许多脱靶事件,而转录自质粒模板的sgRNA没有这样的问题。多重化Digenome-seq可以捕捉到很多被其他方法漏掉的良性脱靶位点。研究人员还在此基础上给出了减少CRISPR-Cas9脱靶效应的指南。
基于整合酶缺陷型慢病毒载体(IDLV)的技术
这一技术可以针对CRISPR和TALENs两种技术:在非同源末端连接(NHEJ)的DNA修复过程中,线性的双链IDLV基因组能够优先整合到双链断裂(DSB)中。根据之前发表的文章,这些IDLV能够引入ZFN处理的细胞,标记DSB的位置。之后,利用线性扩增介导的PCR,可定位IDLV的整合位点,揭示IFN是否击中目标位点。
受到这种方法的启发,美国希望之城贝克曼研究所和浙江大学第一附属医院的研究人员将其修改后应用于CRISPR和TALENs。
研究人员利用表达CRISPR/Cas9核酸酶或TALENs的质粒转染HEK细胞,之后转导了IDLV。这个IDLV携带的基因赋予了抗生素和嘌呤霉素的抗性。核酸酶形成DSB后,允许IDLV整合,使得耐受嘌呤霉素的克隆数量增加了两到三倍。
对于CRISPR/Cas9和TALEN处理过的细胞,研究人员在目标切割位点的60 bp内鉴定出成簇的IDLV整合位点(CLIS)。当他们寻找WAS和TAT基因以外的CLIS时,在TALEN处理的细胞中未找到脱靶位点,而在CRISPR/Cas9核酸酶中发现一些。之后他们利用芯片预测和深度测序来确定IDLV分析的灵敏度,发现它能够检测频率低至1%的脱靶切割。
BLESS
科学家提出一种利用全基因组技术进行DNA双链断裂(DSBs)作图新方法,这种作图法以单核苷酸为基本单位,称之为BLESS(direct in situ breaks labeling, enrichment on streptavidin and next-generation sequencing)。
研究人员利用人类和小鼠细胞以及不同的DNA双链断裂(DSBs)诱导因子和测序平台对BLESS法进行验证。BLESS能够识别端粒末端、SCE核酸内切酶诱导的DNA双链断裂以及复杂的DSB全基因组。作为一种原理证明,我们让人类细胞复制压以敏感性基因组为特点,确定了> 2,000非均匀分布的阿非迪霉素敏感区(ASRS),它们中基因过多,富含微卫星重复序列。人类癌症重排区也富含ASRS,许多癌症相关的基因对复制压具有极高的敏感性。
研究人员称,这种新方法适合各种细胞核实验条件下DSBs全基因组作图,其特异性和分辨率是现在的技术无法实现的。
SITE-Seq
研究人员研发出了一种新生化方法,通过测序(SITE-Seq方法)选择性富集和识别标记基因组DNA末端,从而在纯化的基因组DNA中找到Cas9的切割位点。SITE-Seq是一种生物化学检测方法,因此基因组DNA可以通过一系列sgRNP浓度进行消化,从最低到最高,从而可以进行高分辨率和低切割灵敏度的脱靶位点鉴别,这样研究人员就可以细致全面的检查细胞中可能的脱靶位点,检测突变频率和功能性细胞影响。而且SITE-Seq也能创建高度富集sgRNP切割片段的测序文库,帮助以最小的读取深度进行特异性分析,这是将SITE-Seq作为高通量指导选择工具的一个至关重要的特征。
最终,研究人员利用SITE-Seq来描绘了一组sgRNP生化切割的图谱,检测了靶向人类基因组的sgRNA的Cas9特异性,这些位点的数目取决于sgRNA序列和核酸酶浓度,在较低浓度下鉴定的位点显示出细胞中脱靶突变的倾向性较高,脱靶位点列表也指出细胞内Cas9活性受到sgRNP递送,细胞类型和暴露于核酸酶的持续时间的影响。《Nature Methods》:CRISPR-Cas9全基因组剪切位点图谱
CIRCLE-seq
麻省总医院和哈佛大学的研究人员介绍了一种新的体外筛查方法CIRCLE-seq。他们认为,这种方法在鉴定全基因组的CRISPR/Cas9脱靶突变上,比现有的细胞或生化方法表现更佳。
研究人员认为,这种方法可以鉴定与细胞类型特异的单核苷酸多态性相关的脱靶突变,证明了它有望生成个性化的特异性图谱。同时CIRCLE-seq为鉴定全基因组范围的脱靶突变提供了一种快速全面的方法。
研究人员也检测了新方法的灵敏度。他们获得了针对HBB基因的向导RNA的脱靶结果,并将这个图谱与另一种鉴定方法(Digenome-seq)获得的图谱进行比较。他们发现,CIRCLE-seq鉴定出Digenome-seq所发现的29个脱靶位点中的26个,同时还发现了156个新位点。全面检测CRISPR脱靶效应的新策略
来源:生物通 作者:张迪
为你推荐
 资讯
资讯 新一代 cAMP 偏向型 GLP-1 受体激动剂埃诺格鲁肽(先维盈)获批用于中国成人体重管理
在一项大型III期临床研究中展现出具有临床意义的减重效果:治疗48周时,受试者的平均体重下降达15 4%,其中92 8%的受试者实现了5%以上的体重减轻。
2026-03-06 12:38
 资讯
资讯 11.8亿美元!德琪医药自免TCE双抗ATG-201授权优时比
德琪医药将其自主研发的CD19 CD3双特异性T细胞连接抗体(TCE)ATG-201的全球开发、生产及商业化独家权益授予优时比,用于治疗B细胞相关自身免疫性疾病。
2026-03-04 20:05
 资讯
资讯 君实生物被调出科创50
近日,上海证券交易所,中证指数有限公司联合发布《关于科创50等指数一季度定期调整结果的公告》,本次调整,调入3家,调出3家,其中君实生物被调出科创50指数,此次调整将于202...
2026-03-04 15:30
 资讯
资讯 吉利德比克替拉韦与来那帕韦单片治疗方案在更换抗反转录病毒治疗药物的 HIV 感染者中维持病毒学抑制
新型在研药物组合将全球指南推荐、具有高耐药屏障的整合酶链转移抑制剂比克替拉韦,与同类首创衣壳抑制剂来那帕韦相结合
2026-03-04 13:41
 资讯
资讯 全球首创便携式宫颈无创光动力治疗药物希维她在华获批
CIN2的治疗决策一直是妇科宫颈上皮内病变治疗临床实践中的核心挑战,宫颈环形电切术(LEEP)等切除性手术虽为临床常用的治疗方案,但其可能导致宫颈结构改变、宫颈机能不全,进...
2026-03-04 13:32
 资讯
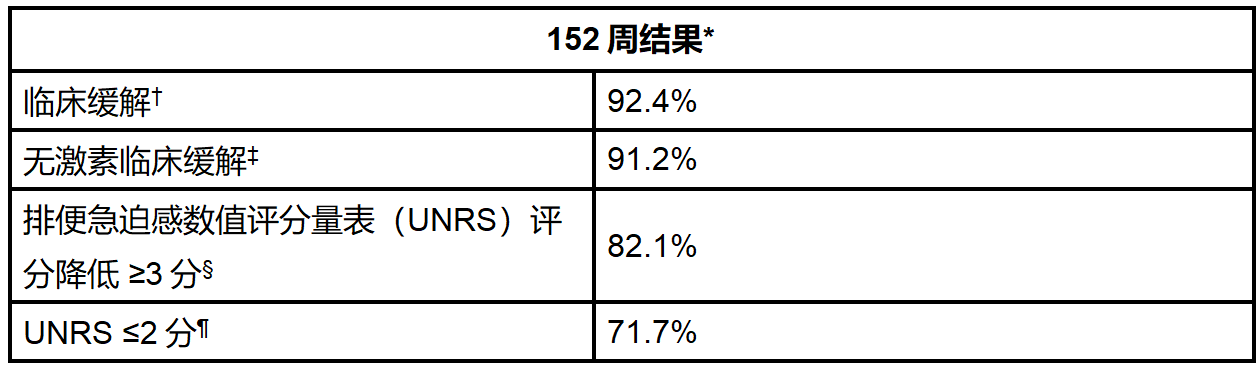
资讯 礼来安妥来利和安妥来(米吉珠单抗)可使克罗恩病患者维持长达三年的无激素缓解
具有里程碑意义的VIVID-2研究数据显示,在治疗后一年实现无激素缓解的患者中,超过90%在三年内仍维持无激素缓解
2026-03-04 13:22
 资讯
资讯 甘李药业一款PROTAC在研药品获批临床试验
3月2日晚间,甘李药业发布公告,在研药品 GLR2037 片的取得国家药监局《药物临床试验批准通知书》。受理号为 CXHL2600098、CXHL2600099,通知书编号为 2026LP00526、2026LP0...
2026-03-03 13:51
 资讯
资讯 非共价(可逆)BTK抑制剂匹妥布替尼获批新适应症
3月2日晚间,信达生物发布公告,公司与礼来中国共同宣布非共价(可逆)BTK抑制剂捷帕力(匹妥布替尼)正式获得中国国家药品监督管理局(NMPA)批准新增适应症,用于治疗既往经过...
2026-03-03 13:19
 资讯
资讯 ICON发布2025年中国生物技术行业调研报告:中国创新领跑全球,95%受访者对未来充满信心
调研显示,中国正成为全球药物创新关键引擎,在细胞疗法、心血管及肿瘤领域持续领跑,AI应用与全球化布局正驱动行业发展
2026-03-03 10:58
 资讯
资讯 国家卫健委:做好《生物医学新技术临床研究和临床转化应用管理条例》贯彻落实
近日,国家卫健委科教司发布《做好 贯彻落实,高质量满足人民群众健康需求,支撑生物医药产业更高水平发展》,表示,距离《条例》正式施行已不足三个月,相关配套文件正在加紧...
2026-03-02 14:23
 资讯
资讯 百济神州的业绩
2月27日,百济神州发布2025年业绩快报,报告期内,公司产品收入为377 70亿元,较上年同比上升39 9%,上年同期产品收入为269 94亿元,公司营业总收入为382 05亿元,较上年同...
2026-03-01 23:03
 资讯
资讯 国家卫健委启动为期3年的卫生健康领域科研诚信专项治理
近日,国家卫生健康委印发《加强医学科研诚信专项治理的工作方案》,启动实施为期3年的卫生健康领域科研诚信专项治理。
2026-03-01 17:57
 资讯
资讯 新版《人体生物监测质量保证规范》国家标准明日实施(附全文)
市场监管总局(国家标准委)批准发布了《人体生物监测质量保证规范》国家标准,该标准由国家疾病预防控制局组织起草,自3月1日起正式实施。
2026-02-28 14:04








