来自斯坦福大学、加州大学旧金山分校的研究人员报告称,他们利用CRISPR干扰(CRISPRi)技术,对枯草杆菌的必需基因进行了全面的功能分析。这项研究发布在5月26日的《细胞》(Cell)杂志上。
斯坦福大学的齐磊(Lei S. Qi)博士、Kerwyn Casey Huang博士,以及加州大学旧金山分校的Carol A. Gross博士是这篇论文的共同通讯作者。齐磊早年毕业于清华大学,当前的主要研究方向是应用基因组工程和CRISPR技术研究与细胞分化、增殖、表观遗 传调控和疾病相关的遗传互作网络。已经在Cell、Nature Biotechnology、Nature Methods等杂志上接连发表了多篇CRISPR文章。
2013年,齐磊博士在Cell发布论文介绍了课题组开发的基因沉默技术。齐磊领导研究人员将CRISPR用于基因表达序列特异性调控的平台,研发 出了一种基于Cas9的基因调控方法。他们将这个系统称为CRISPRi,并指出这种RNA导向的DNA识别平台是基因组范围内基因表达选择性抑制的一种 简单的新方法。
2015年1月,齐磊与加州大学旧金山分校的Wendell A. Lim合作,开发了一种CRISPR支架RNA(scRNA)。scRNA编码了靶位点和调控功能,能同时实现对不同基因的激活和抑制。这项研究发表在 Cell杂志上。这项研究为设计基因表达程序的研究者们提供了一个有力工具。
2015年,齐磊博士与清华大学自动化系的汪小我课题组合作,根据已发表的研究总结的一套sgRNA设计规则,开发出了一种综合性的计算工具。他们 报道了一种全基因组sgRNA设计工具,并提供了一个在线网站,用于预测高效而特异的sgRNA。他们将这一工具命名为CRISPR-ERA,可用于 CRISPR介导的基因编辑、抑制和激活。
在这篇Cell文章中,齐磊和合着作者们指出必需基因发挥功能支持了核心的细胞过程。调查必需基因功能之间的关系对于认识控制细菌生 长的机制,以及推动药物开发至关重要。然而,当前只有少数几种方法可用来在体内评估必需基因功能阐明它们的关系。由于必需基因丧失功能会导致细胞无法生 存,无论是基因缺失文库还是饱和转座子诱变都不能用来研究必需基因。有几种高通量方法已被用于鉴别或扰乱真核生物中的必需基因,包括使得mRNAs 3′UTR失稳,CRISPR/Cas9基因编辑,和CRISPR/dCas9转录调控技术。当前唯一在细菌中筛查必需基因的研究利用了反义RNA敲低来 筛查抗生素敏感性,由于效率多变这一方法的功用受到限制。
在新研究中,研究人员利用CRISPRi来敲低枯草杆菌中每个必需基因,探究了它们的表型。他们利用化学基因组学建立的高可信度必需基因网络显示了 关系遥远的一些过程之间广泛的相互联系,确定了一些未确定特征的抗生素的作用模式。重要地是,研究人员发现轻微敲低必需基因功能可显着降低静止期存活而不 影响最高生长速率。最后,高通量显微镜分析表明,细胞形态对于轻微敲低相对不敏感,而受到删除基因功能的显着影响,揭示了细胞生长与形态之间的密切联系。
新研究为在体内系统调查必需基因功能提供了一个广泛适用于各种微生物和比较分析的一个框架。
来源:生物通 作者:何嫱
为你推荐
 资讯
资讯 悦唯医疗完成近亿元A++轮投资,加速重症冠心病诊疗全流程创新器械研发与国产替代
此次融资将主要用于深化冠心病诊疗全流程创新器械和脉动式左心室辅助系统等新产品的研发,以及加速已获准上市的心脏稳定器等产品的市场推广。
2025-04-03 09:28
 资讯
资讯 海尔盈康一生启动孤独症儿童关爱行动,创新罕见病可持续公益新生态
本次活动聚焦孤独症儿童的诊疗,探讨交流AI赋能全流程防治康体系创新、前沿性生物科技诊疗技术等话题,旨在通过生态联盟的力量推动医学研究、科技创新与人文关怀的融合,让“星...
2025-04-03 09:11
 资讯
资讯 《NPJ digital medicine》刊发李冬梅教授团队成果:AI赋能高效识别眼睑肿物
亚太眼整形外科学会主席、中华医学会眼科分会眼整形眼眶病学组副组长李冬梅教授团队携手爱尔数字眼科研究所,在《NPJ digital medicine》(影响因子:12 4)学术期刊发表团队...
文/李林 2025-04-02 10:27
 资讯
资讯 默克全球执行副总裁周虹:合作与创新是默克未来五年战略的两大关键词
近日,德国默克医药健康全球执行副总裁、中国及国际市场负责人周虹带领医药健康中国及国际市场管理团队开启了2025年度首次“中国行”。
2025-04-01 17:11
 资讯
资讯 首个且唯一,阿斯利康PD-L1单抗获FDA批准治疗肌层浸润性膀胱癌
度伐利尤单抗联合吉西他滨和顺铂作为新辅助治疗,随后度伐利尤单抗作为根治性膀胱切除术后的辅助单药治疗,用于治疗肌层浸润性膀胱癌成年患者。
2025-04-01 14:37
 资讯
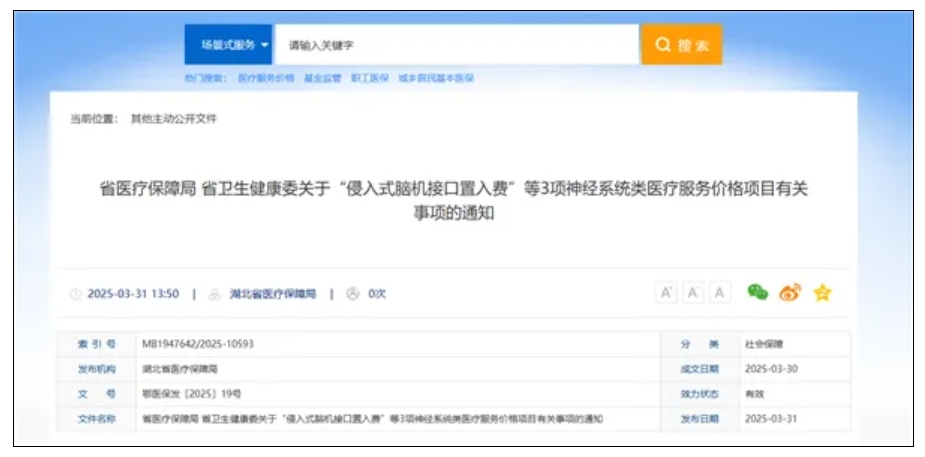
资讯 全国首个,湖北为脑机接口医疗服务定价
昨日(3月31日),据“湖北发布”消息,湖北省医保局发布全国首个脑机接口医疗服务价格,其中,侵入式脑机接口置入费6552元 次,侵入式脑机接口取出费3139元 次,非侵入式脑机...
2025-04-01 11:03
 资讯
资讯 一款国产创新流感药,获批
近日,据国家药监局官网信息显示,青峰医药下属子公司江西科睿药自主研发的1类创新药玛舒拉沙韦片(商品名:伊速达)正式获批上市,用于既往健康的12岁及以上青少年和成人单纯性...
2025-04-01 10:22
 资讯
资讯 26省联盟药品集采启动,聚焦妇科用药和造影剂
近日,山西省药械集中招标采购中心发布《关于做好二十六省联盟药品集中带量采购品种数据填报工作的通知》,开展相关采购数据填报工作。
2025-03-31 21:48
 资讯
资讯 优时比罗泽利昔珠单抗注射液(优迪革)中国获批,全球首个且唯一双亚型创新药治疗全身型重症肌无力
作为唯一人源化、高亲和力且具备创新修饰结构的IgG4单抗,关键Ⅲ期MycarinG试验证实罗泽利昔珠单抗注射液(优迪革®)较安慰剂显著改善全身型重症肌无力患者的多个临床终点与结局。
2025-03-31 15:58
 资讯
资讯 从手术麻醉到生命全周期护航,麻醉学科发展拓宽生命边界
3月26日,由中华医学会麻醉学分会、中国医师协会麻醉学医师分会等23家学协会共同举办的2025年中国麻醉周学术活动的启动仪式举办,该活动以“生命之重,大医精诚——守生命保驾护...
2025-03-31 15:30
 资讯
资讯 欧狄沃联合逸沃成为中国目前唯一获批的肝细胞癌一线双免疫联合疗法
欧狄沃联合逸沃对比仑伐替尼或索拉非尼,可显著改善不可切除肝细胞癌一线患者的总生存期(OS),客观缓解率(ORR)可改善近3倍,中位缓解持续时间(mDOR)达30个月
2025-03-31 13:45
 资讯
资讯 罗氏制药榜首 “现金牛” 产品罗可适(奥瑞利珠单抗)在华获批:开启多发性硬化症一年两次治疗新时代
罗氏制药今日(3月31日)宣布,其旗下创新药罗可适®(Ocrevus®,通用名:奥瑞利珠单抗注射液 ocrelizumab injection)正式获得中国国家药品监督管理局批准,每六个月静脉输...
2025-03-31 13:39
 资讯
资讯 三生有幸,医者仁心:三生制药向全体医药工作者致敬!
3月30日是国际医师节,由三生制药公益支持的以“三生有幸,医者仁心”为主题的公益活动,携手20位医生代表,以寄语海报的形式,共同向全体医护人员表达诚挚的祝福与关爱。
2025-03-30 17:38
 资讯
资讯 新版药典自2025年10月1日起实施
3月25日,国家药监局官网发布《国家药监局 国家卫生健康委关于颁布2025年版的公告(2025年第29号)》,2025年版《中国药典》自2025年10月1日起施行。
2025-03-30 17:07