美国国家儿童医院(Nationwide Children's Hospital)的研发人员最近在Genome Biology上发布了一个自主开发的分析软件,表示这个软件使寻找全基因组致病变异从几周缩短到按几十个小时。
第一个人类基因组测序完成耗时大约13年,耗费30亿美元,而现在技术测序技术的发展,使得即使是很小的研究小组都可以在几天之内完成基因组测序。但是从测序产生的巨大的数据分析得出真正能用于研究或者临床的信息一直是一个挑战。而彼得·怀特博士和他带领的团队针对这个问题,利用新颖的计算技术,开发了一个名为“丘吉尔”(Churchill)的计算管道,表示“丘吉尔”可以在短短90分钟内完成全基因组样品的有效分析。
“丘吉尔”自动输入原始序列资料,通过一系列密集复杂和计算,最终分析出有临床或者科研意义的的遗传变异体。这个过程中的每一步,“丘吉尔”都有优化,以显着减少分析时间,但不损害数据的完整性,该分析是100%的可重复性。“丘吉尔”采用的平行化(parallelization)的算法克服了染色体带来的平行化限制,极大提升了数据输入的平衡性和分析中数据重新组合,去分,再校准和基因型分型的执行性。通过检查在数据分析过程中的计算资源的利用,相比其他两种分析管道-HugeSeq和GATK-Queue只能分别利用46%和30%的数据资源,“丘吉尔”的利用率达到了92%,并在多个服务器非常有效地进行缩放。“丘吉尔”输出结果,在与其他计算管道比较,被证明具有最高99.7%的灵敏度;最高99.99%的精读和99.66%最高整体诊断效率。
这种效率和能力,证明“丘吉尔”或能够进行人口规模的基因组分析。为了证明“丘吉尔”的能力,怀特博士和他的团队成功地分析了千人基因组项目所产生的第一阶段的原始数据(千人基因组项目是以生成世界各地的多个群体人类遗传变异的公众目录为目的的国际合作项目)。利用亚马逊网络服务(AWS)的云计算资源,“丘吉尔”仅用七天便完成1088个全基因组样本的分析,并确定了数以百万计的新的遗传变异。
“丘吉尔”的发布是测序技术一个极大的进步。它极大降低测序分析的成本,突破了当今测序分析计算的瓶颈,特别为现在大人口规模的基因组学的研究提供便利。
来源:生物谷
为你推荐
 资讯
资讯 带状疱疹疫苗“遇冷”,百克生物2024年净利润腰斩
近日,国内疫苗龙头企业百克生物发布2024年年报,数据显示,其报告期内实现营收12 29亿元,同比下降32 64%;归属于上市公司股东的净利润2 32亿元,同比下降53 67%。对于营收...
2025-04-23 12:59
 资讯
资讯 重庆常用药联盟接续集采中选结果
近日,重庆常用药联盟接续集采中选情况公布,该联盟由重庆牵头,联合湖北、广西、海南、贵州、云南、青海、宁夏、新疆及新疆生产建设兵团等十省(区、市)开展的药品集中带量采...
2025-04-21 18:48
 资讯
资讯 全周期智控慢病,诺和诺德与京东健康开启战略合作
2025年4月21日,全球领先的生物制药公司诺和诺德与京东健康在北京正式签署战略合作协议,标志着双方在糖尿病和体重管理领域的合作进入新阶段。依托诺和诺德百年深耕慢病领域的专...
2025-04-21 15:57
 资讯
资讯 康方生物1类新药依若奇单抗上市申请获批,用于中重度斑块状银屑病成人患者
该药是我国第一个且唯一获批上市的IL-12 IL-23“双靶向”单克隆抗体新药,是康方生物自身免疫性疾病领域首个获批上市的一类新药。
2025-04-21 13:39
 资讯
资讯 阿斯利康乳腺癌1类创新药卡匹色替片中国获批
该药适用于联合氟维司群用于转移性阶段至少接受过一种内分泌治疗后疾病进展,或在辅助治疗期间或完成辅助治疗后12个月内复发的激素受体(HR)阳性、人表皮生长因子受体2(HER2)...
2025-04-21 11:02
 资讯
资讯 辉瑞宣布终止一款口服GLP-1减肥药的临床开发
近日,辉瑞在其官网宣布,决定终止开发口服胰高血糖素样肽-1受体(GLP-1R)激动剂Danuglipron(PF-06882961),原因系在一项有关用药剂量的临床试验中,一名患者出现了可能由该...
2025-04-21 10:29
 资讯
资讯 福建省医保局印发单列门诊统筹支付医保药品目录(2024年版)
根据2024年6月发布的《福建省医保药品单列门诊统筹支付管理办法(试行)》,为了让参保患者无需住院、在门诊就医也能用上国家谈判药品、享受医保待遇,将适用于门诊治疗、使用周...
2025-04-20 13:34
 资讯
资讯 首批中国消费名品名单,医药健康企业有哪些?
近日,工业和信息化部办公厅发布首批中国消费名品名单,分为中国消费名品名单和中国消费名品成长企业名单。首批中国消费名品名单共包括93个企业品牌和43个区域品牌。中国消费名...
2025-04-20 11:17
 资讯
资讯 携手共绘“个性化近视手术”新蓝图:爱尔眼科与爱尔康启动100家医院全光塑技术战略合作
双方将以技术共享为核心,以人才培养为支撑,以科研协作为纽带,全力推进屈光手术标准化诊疗体系建设,加速前沿技术在临床领域的普及应用
文/ 屈慧莹 2025-04-19 23:35
 资讯
资讯 CDE:简化港澳已上市传统口服中成药内地上市注册审批申报资料及技术要求
允许香港、澳门特区本地登记的生产企业持有,并经香港、澳门特区药品监督管理部门批准上市且在香港、澳门特区使用15年以上,生产过程符合药品生产质量管理规范(GMP)要求的传统...
2025-04-18 18:54
 资讯
资讯 君德医药完成近亿元A轮融资,加速推进创新药械组合平台建设与产品上市
本轮融资主要用于首个减重口服器械的注册及生产销售,以及加速多个核心创新药械组合技术平台的产品管线研发进程。
2025-04-18 14:34
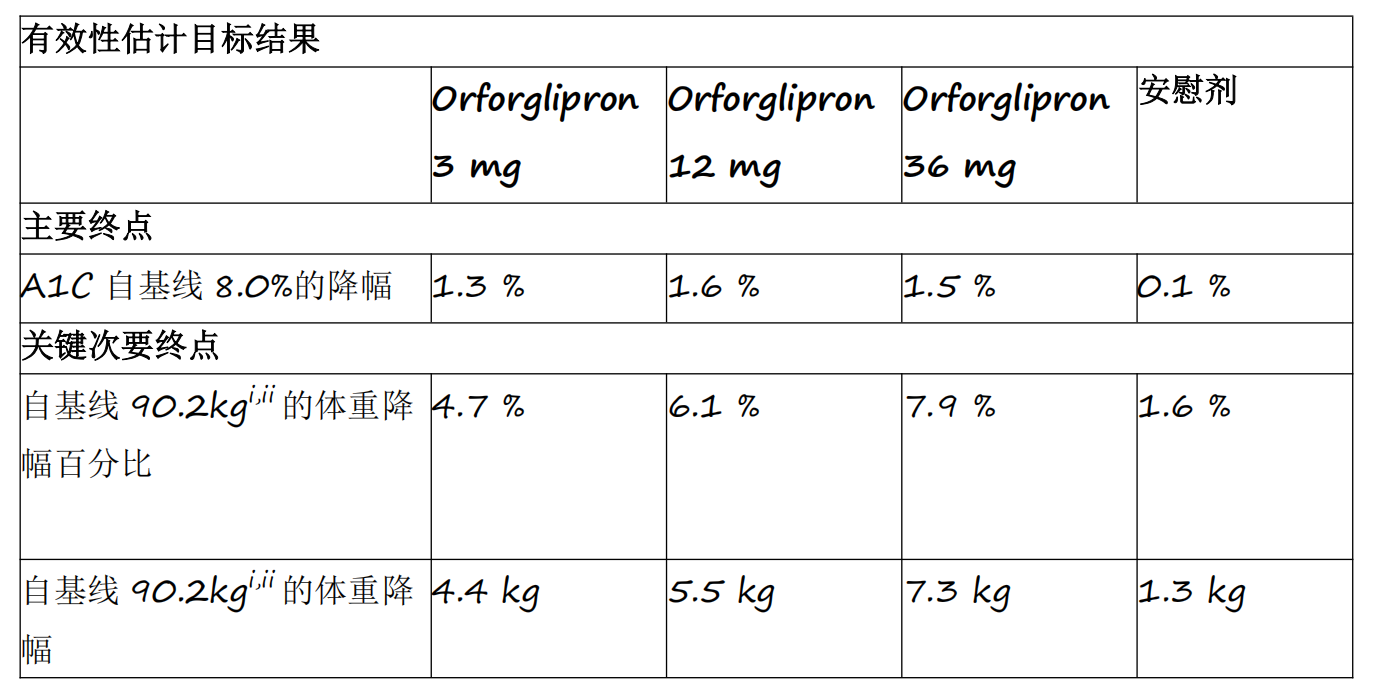 资讯
资讯 礼来首个小分子口服GLP-1RA药物orforglipron 3期临床研究成功
Orforglipron是首个成功完成3期临床研究的小分子GLP-1类药物,各剂量组平均A1C降幅为1 3%至1 6%
2025-04-18 14:12